CG Exercise #04
Visualisierung √úbungsblatt #02
Computergraphik √úbungsblatt #04
OpenGL Tutorial: Rendering Primitives
OpenGL: Erstes eigenes Programm
Aufgabe 4.1: Linien zeichnen (CMake)
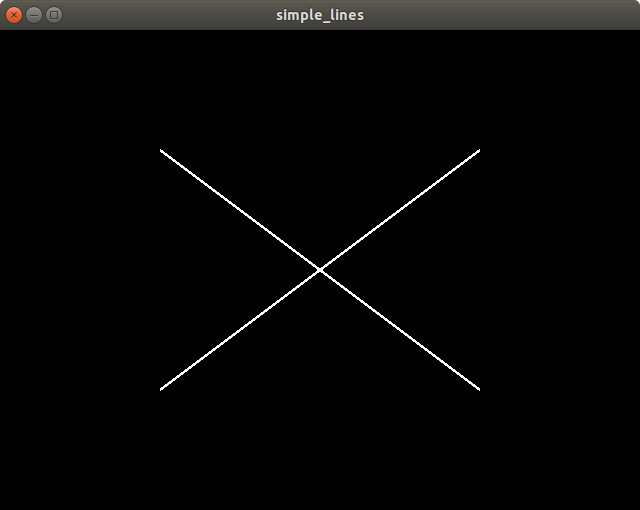
Linien mit OpenGL
√Ėffnen Sie ein Linux-Terminal Fenster und navigieren Sie mittels cd in das “qt_template” Verzeichnis des glVertex Frameworks, welches Sie bereits zu Anfang der √úbungen installiert haben.
Unter Linux: √úbersetzen Sie die Programmiervorlage und starten Sie sie:
cmake . && make && ./qt_template
Auf Windows und Mac: Verwenden Sie QtCreator wie in der nächsten Aufgabe.
Sie sollten nun ein (fast) leeres Fenster sehen. Dies ist der Startpunkt f√ľr die ersten OpenGL Befehle im ersten eigenen OpenGL Programm:
- Verwenden Sie einen Plain-Text-Editor wie den Emacs oder Visual Studio Code, um das C++ Programm qt_template.cpp im entsprechenden Unterverzeichnis zu verändern.
- Zeichnen Sie eine einzelne Linie, wie in der Vorlage bereits als Kommentar vorgeschlagen.
- Zeichnen Sie eine weitere Linie, so dass ein Kreuz entsteht.
- √Ąndern Sie die Hintergrundfarbe auf Himmelblau.
- Entlang welcher Achse schaut der Betrachter?
- Welche zwei Achsen definieren die Bildebene?
Materialien:
- OpenGL Framework: glVertex
- Vorlesung: GL Vertices & Attribute
- LGL: OpenGL Cheat Sheet
- LGL Dokumentation: Die Aufrufsyntax aller Framework-Funktionen mit dem Prefix lgl wie z.B. lglRender() ist in der Klassendokumentation nachzulesen. Dazu √∂ffnen Sie mit Firefox im Verzeichnis “docs/html” den Dokumentationsindex “index.html” und browsen die Dokumentation der Datei “glvertex_api.h”.
- OpenGL 1.2 Programming Guide
Aufgabe 4.2: Dreiecke zeichnen (Qt Creator)
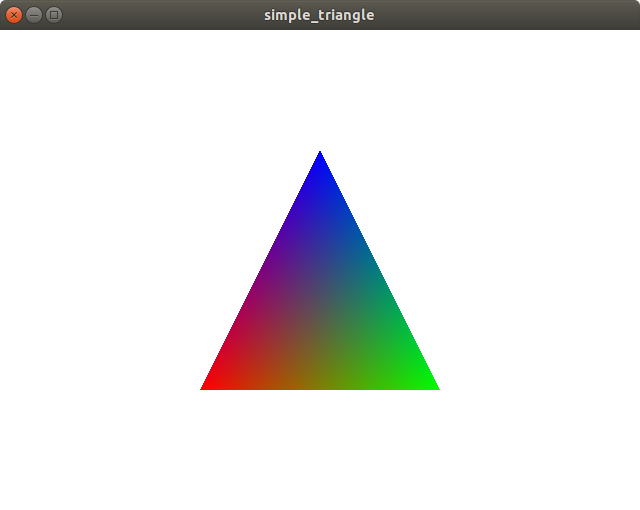
Farbige Dreiecke mit OpenGL
Auf Linux, Windows und Mac: √Ėffnen Sie nun dasselbe Programm nicht vom Linux-Terminal aus, sondern mit Hilfe von QtCreator. Dazu doppel-klicken Sie auf die .pro-Datei im Verzeichnis “qt_template”. F√ľhren Sie das Programm nun in QtCreator aus, indem Sie auf den gr√ľnen Pfeil klicken.
Sie sollten nun dieselbe Darstellung wie in der vorhergehenden Aufgabe sehen, und k√∂nnen weitere OpenGL Befehle hinzuf√ľgen:
- Zeichnen Sie wie im OpenGL Cheat Sheet beschrieben ein weißes Dreieck (siehe auch Vorlesung).
- Zeichnen Sie wie im OpenGL Cheat Sheet beschrieben ein farbiges Dreieck mit unterschiedlichen Farb-Attributen f√ľr jeden Vertex.
- Zeichnen Sie ein weiteres Dreieck, welches sich mit dem ersten schneidet.
Hinweis: QtCreator ist die empfohlene Entwicklungsumgebung f√ľr Linux und Windows, man kann aber auch z.B. auf Visual-Studio Code oder die Kommandozeile (via CMake & Emacs) wie in der vorherigen Aufgabe ausweichen.

Blend File
CC-BY DarkLimits Studios
modified for Eevee
by A. Kalisz
Aufgabe 4.3: Blender Geometrie laden
| Teil | Videos des WS 2021/22 | Thema |
|---|---|---|
| OBJs | YT | Geometrie importieren bzw laden |
Es w√§re doch sch√∂n, ein eigenes graphisches Objekt f√ľr OpenGL zu verwenden anstelle von langweiligen Dreiecken. Dazu k√∂nnen Sie Ihr aktuelles Objekt in Blender im Alias WaveFront-Format (.obj) exportieren.
Beim Export sollten die folgenden Optionen ausgewählt sein:
- Faces/Mesh triangulieren
- Normalen schreiben
- UV-Koordinaten schreiben
- Modfier anwenden (z.B. Subdivision Surface)
Beim Export sollten die folgenden Optionen NICHT ausgewählt sein:
- Materialeigenschaften schreiben
- Nur ausgewähltes Objekt schreiben
Laden Sie nun Ihr eigenes exportiertes Objekt mit der OpenGL-Programmiervorlage im Verzeichnis “qt_template”. Alternativ k√∂nnen Sie auch die Obj-Datei des oben abgebildeten Elefanten verwenden. Ihre exportierte Obj-Datei muss im selben Verzeichnis liegen. Dann laden Sie die Obj-Datei mit lglLoadObj(“filename”) in initializeOpenGL():
vbo = lglLoadObj("teapot.obj");
Dazu benötigen Sie eine entsprechende Instanzvariable (engl. member variable) vom Typ lglVBO* mit Namen vbo.
Testen Sie nun, ob die Datei korrekt geladen wurde, indem Sie √ľberpr√ľfen, ob die Instanzvariable einen NULL Zeiger enth√§lt und brechen Sie das Programm in diesem Fall ab → exit(1).
Geben Sie weiterhin die Abmessungen Ihres Objektes aus, welche Sie mit der Methode lglGetExtent() erhalten (Ausgabe via operator <<).
Das geladene Objekt wird nun mit der Methode lglRender(vbo) in renderOpenGL() gezeichnet.
Es kann sein, dass die Abmessungen des Objektes zu gro√ü sind, so dass Sie es erst skalieren m√ľssen, um es sinnvoll rendern zu k√∂nnen. Verwenden Sie dazu den Befehl lglScale() in initializeOpenGL(). Der Teapot (Datei “teapot.obj”) muss z.B. mit dem Faktor 0.01 skaliert werden.
Aufgabe 4.4: OpenGL State Machine
Verändern Sie die Darstellung, indem Sie verschiedene Effekte durch Parametereinstellungen der OpenGL State Machine anschalten:
Orthographische Projektion:
Ihr Objekt wird noch verzerrt dargestellt, weil keine Projektion definiert ist. Verwenden Sie die folgende orthographische Projektion analog zu glOrtho (siehe auch OpenGL Cheat Sheet und OpenGL Programming Guide):
lglProjection(-1, 1, -1, 1, -1, 1)
Ihr Objekt wird nun immer noch verzerrt dargestellt, weil das Seitenverh√§ltnis (aspect ratio) noch nicht ber√ľcksichtigt wird. Das Seitenverh√§ltnis berechnet sich zu aspect = (float)width()/height(). An welchen zwei Stellen in obiger Projektion muss das Seitenverh√§ltnis anstelle einer 1 verwendet werden?
Z-Buffer:
Schalten Sie testweise den Z-Buffer aus. Welchen lgl-Befehl bzw. welche Taste benötigen Sie dazu?
Optional: Wireframe Darstellung:
Stellen Sie Ihr Objekt im sog. Wireframe-Modus dar. Welchen lgl-Befehl benötigen Sie dazu?
Optional: Obj-Datei animieren:
Animieren Sie Ihr Objekt, indem Sie es rotieren lassen. Verwenden Sie dazu den Befehl lglRotate(). Um welchen Winkel $\Delta\alpha=\omega \cdot \Delta t$ m√ľssen Sie das Objekt in jeden Frame rotieren, wenn Sie 30 fps annehmen und eine volle Umdrehung alle 3 Sekunden stattfinden soll?
Hausaufgaben bis zum nächsten Praktikum
Musterlösung: Bitte stellen Sie bitte bei Bedarf Ihre Lösung aus dem Praktikum kurz vor, anhand derer wir gemeinsam die Musterlösung entwickeln können.
1. Matrizen:
- Rotation und Translation:
In der Vorlesung haben wir beispielhaft mit einer Transformationsmatrix einen 3D Vektor transformiert. Die Transformation bestand aus einer Rotation und einer nachfolgenden Translation. Wie nennen wir so eine Transformation? - Wie sieht diese Matrix konkret aus, d.h. aus welchen Teilmatrizen ist diese aufgebaut? Erklären Sie diese Bestandteile der Matrix!
- Berechnen Sie die 4×4 Modellierungs-Matrix $M_M$, welche eine Rotation um 180 Grad um die Y-Achse und eine anschlie√üende Translation um den Vektor (0,0,−5) repr√§sentiert.
- Viewing-Transformation:
Berechnen Sie die View-Matrix $M_V$ einer Kamera, die an der Position (0,0,−10) steht und entlang der positiven Z-Achse schaut. Hinweis: Die Viewing-Transformation ist die Inverse der Modellierungstransformation der Kamera! Standardm√§√üig schaut die Kamera in OpenGL entlang der negativen Z-Achse!
2. Animation:
- Zeichnen Sie mit dem OpenGL-Framework einen Einheitsw√ľrfel, der aus 6 verschiedenfarbigen
LGL_QUADs besteht. - Als nächstes soll dieser mit der Winkelgeschwindigkeit $\omega=30$ rotieren. Verwenden Sie den Funktionsparameter
dtals $\Delta t$. Eine Objektrotation erreicht man mit lglRotate.
3. Optionale Animationen:
- Verschieben Sie Ihr Objekt, indem Sie es mit einer H√∂he $h$ translieren (mittels lglTranslate). Achtung: vergessen Sie nicht, die Verschiebung des letzten Frames am Anfang des n√§chsten Frames mit lglLoadIdentity() wieder r√ľckg√§ngig zu machen.
- Versuchen Sie, Ihr Objekt zus√§tzlich zur Rotation h√ľpfen zu lassen, indem Sie mit der H√∂he $h$ translieren und die H√∂he $h$ und die Fallgeschwindigkeit $v$ in jedem Frame anpassen:
- $v := v+G\Delta t$ mit $G=-\frac{1}{6}9.81\frac{m}{s^2}$ (Mond-Gravitation)
- $h := h+v\Delta t$
- $v := -v$ f√ľr $h<0$

